 Cryptogamie, Algologie
31 (4) - Pages 451-465
Cryptogamie, Algologie
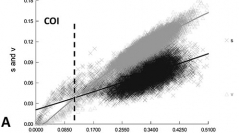
31 (4) - Pages 451-465Le code-barres mitochondrial ADN COI, un segment partiel du gène rARN LSU et le marqueur plastidial rARN UPA, ont été séquencés sur un total de 290 échantillons de Florideophytes (représentant 17 ordres) issus du projet "Hawaiian Rhodophyta Biodiversity Survey". Le marqueur COI est globalement plus riche en A-T (> 60 ) comparé aux deux autres marqueurs rARN, mais aussi le moins conservé, avec la troisième position des codons présentant une forte substitution. Sur la base d'un modèle F84, la saturation est atteinte à une distance d'environ 0,11 pour COI and 0,30 pour LSU, mais absente pour UPA, et certaines différences existent quand les ordres ou groupes de familles sont examinés individuellement. Le taux de réussite du séquençage pour les trois marqueurs varient de 46,8 pour COI, 64,7 pour UPA et 79,6 pour LSU, indiquant des différences substantielles pour l'obtention de données. La concaténation successive des marqueurs, en démarrant du moins saturé (UPA), et en ajoutant LSU et COI par ordre de saturation (UPA LSU, puis UPA LSU COI) conduit à une augmentation forte du soutien bootstrap par la méthode d'analyse du neighbor-joining, indiquant l'utilité phylogénétique de séquences courtes d'ADN ("barcode-like") obtenues dans le cadre de projets d'exploration de la biodiversité.
Également disponible sur Connect.barcodeoflife